Plateforme d’Analyse Transcriptomique de cellule individuelle par DROP-SEQ
Financée depuis 2018
But
L’infrastructure scGenAP vise à générer et partager les données de génomiques produites suite à l’isolation de cellules individuelles de la rétine et le séquençage de leurs transcriptomes. Cette approche, mieux connue sous le terme anglais de « Single-cell RNA sequencing » (abrégé scRNAseq) fut perfectionnée dans notre laboratoire en collaboration avec le Dr. Andelfinger, au Centre de Recherche du CHU Sainte-Justine.
Description
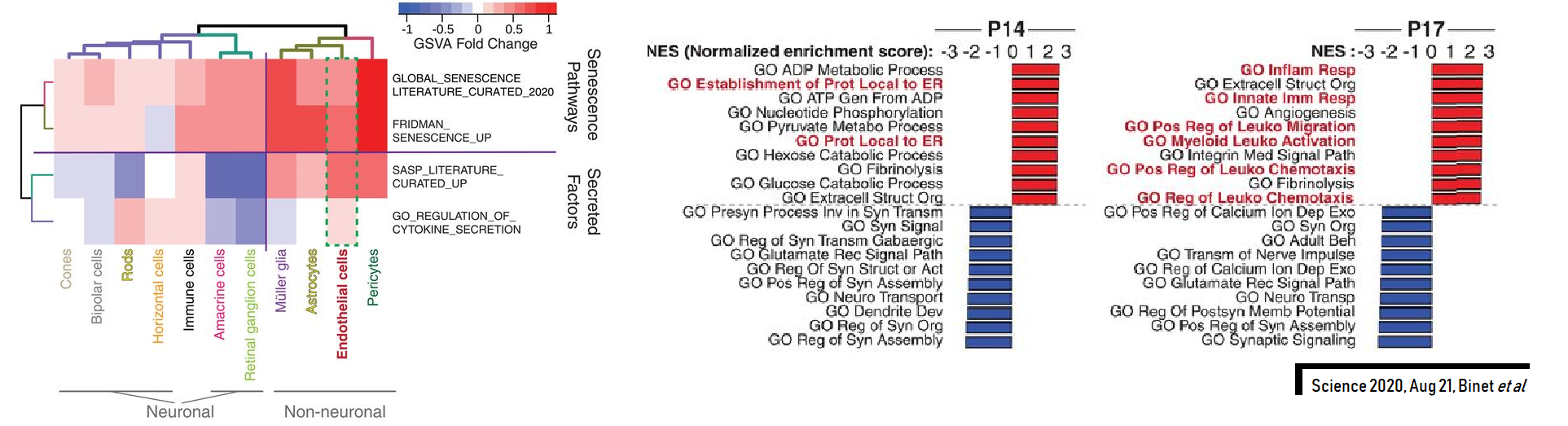
La technologie de scRNAseq nous permet d’évaluer le transcriptome (mRNA polyA) de chaque cellule vivante préalablement dissociée de rétines (ou autre organe d’intérêt), à différents stades de développement, tant dans un contexte physiologique que pathologique, et ce peu importe l’espèce dont le génome est disponible (afin de pouvoir aligner les données de séquençage).
Les données disponibles à l’heure actuelle proviennent d’analyse faites sur des rétines de souris âgées de 5 à 17 jours, en condition normale ou de rétinopathies (OIR). Les souris analysées proviennent de lignées aux origines génétiques différentes (C57B6 WT et S129 WT). D’autre analyses sont prévues à des âges et origines différents et selon des protocoles de dissociation et d’enrichissement cellulaires ciblés. De plus, l’analyse de rétines fœtales humaines est en cours.
Impact
Comparée au RNAseq classique (« bulk » RNAseq), la technologie de scRNAseq permet d’évaluer les variations transcriptomiques intra-échantillon qui sont dues à l’hétérogénéité cellulaire propre aux tissues vivants. Cette technologie est donc en rupture avec la technologie de RNAseq tant au niveau de l’analyse transcriptomique que de l’interprétation de la variabilité cellulaire au sein d’un échantillon, et devient rapidement l’outil prédominant pour l’étude de l’expression génique tissulaire.
Accessibilité
Cette infrastructure est disponible pour tous les chercheurs membres du RRSV. En effet, il est important de favoriser l’accès et l’utilisation de données publiées ou en voie de l’être afin de favoriser des collaborations au sein du Réseau et permettre à plusieurs laboratoires de tirer profit de la technologie de scRNAseq. Cet avantage compétitif permet aux chercheurs de tester leurs hypothèses in sillico pour la soumission de demandes de financement et d’étoffer leurs manuscrits en faisant usage de données bio-informatiques existantes. De nouveaux modèles pourraient être analysés par scRNAseq pour des projets collaboratifs de plus longues envergures.
Nous mettons en place un site Web donnant un accès privilégié aux membres du Réseau à nos données de scRNAseq publiées (accès public) ou en voie de l’être (accès privé). Chaque PI pourra choisir de donner accès à une banque de données en voie de publication dans le contexte de collaboration. Nous croyons qu’un meilleur partage des données pourrait favoriser de plus étroites collaborations au sein de notre Réseau.
Responsable
Jean-Sébastien Joyal, MD, FRCPC, PhD, Professeur adjoint, Département de Pédiatrie, Professeur accrédité, Département de pharmacologie, Université de Montréal; Professeur associé, Département de pharmacologie et de thérapeutique, Université McGill, Pédiatre-intensiviste, CHU Sainte-Justine
Personne contact
Gael Cagnone – singlecellacademy@gmail.com
Source de financement
Réseau de cherche en santé de la vision